La epidemia de coronavirus COVID-19 tiene un origen natural.

Declaración de la UNESCO sobre el Covid-19: consideraciones éticas desde una perspectiva global.
01/05/2020
Los principales centros de investigación biomédica en España permanecen cerrados desde hace más de un mes.
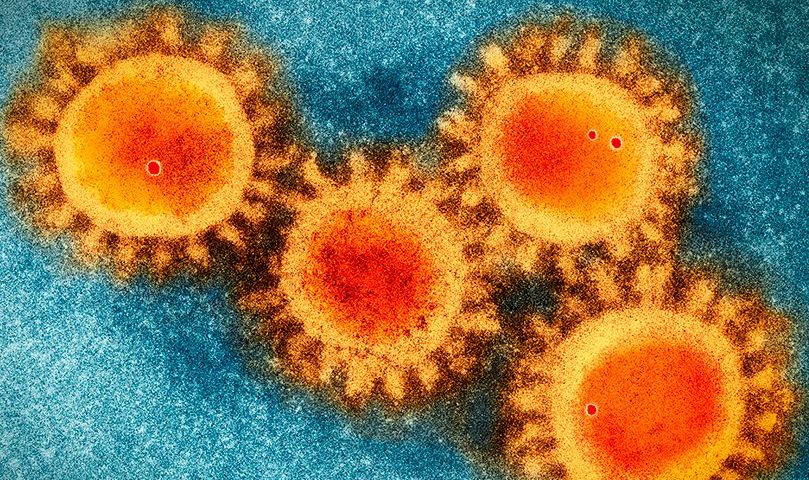
04/05/2020Publicado en Scripps Research, el 17 de marzo de 2020 (imagen de portada, Microscopía electrónica del coronavirus COVID-19.virus).
El nuevo coronavirus SARS-CoV-2 que surgió en la ciudad de Wuhan, China, el año pasado y desde entonces ha causado una epidemia COVID-19 a gran escala y se extendió a más de 70 países, es el producto de la evolución natural, según los hallazgos publicados el 17 de marzo en la revista Nature Medicine. [El SARS-CoV-2 es el séptimo coronavirus conocido que infecta a los seres humanos; El SARS-CoV, el MERS-CoV y el SARS-CoV-2 pueden causar enfermedades graves, mientras que HKU1, NL63, OC43 y 229E se asocian con síntomas leves. Aquí revisamos lo que se puede deducir sobre el origen del SARS-CoV-2 a partir del análisis comparativo de los datos genómicos. Ofrecemos una perspectiva sobre las características notables del genoma del SARS-CoV-2 y discutimos los escenarios por los que podrían haber surgido. Nuestros análisis muestran claramente que SARS-CoV-2 no es una construcción de laboratorio o un virus manipulado intencionalmente SARS-CoV-2.]
El análisis de los datos públicos de las secuencias del SARS-CoV-2 y los virus relacionados no muestra evidencia de que el virus se haya diseñado en un laboratorio. De acuerdo con Kristian Andersen, PhD, profesor asociado de inmunología y microbiología en Scripps Research y autor correspondiente del trabajo publicado «Al comparar los datos de secuencia del genoma disponibles para cepas coronavirus conocidas, podemos determinar firmemente que el SARS-CoV-2 se originó a través de procesos naturales«. Además de Andersen, otros autores del artículo, «El origen proximal de SARS-CoV-2«, son Robert F. Garry, de la Universidad de Tulane; Edward Holmes, de la Universidad de Sydney; Andrew Rambaut, de la Universidad de Edinburgh; W. Ian Lipkin, de la Universidad de Columbia.
Los coronavirus son una gran familia de virus que pueden causar enfermedades que varían ampliamente en gravedad. La primera enfermedad grave conocida causada por un coronavirus surgió con la epidemia de Síndrome Respiratorio Agudo Severo (SARS) de 2003 en China. Un segundo brote de enfermedad grave comenzó en 2012 en Arabia Saudí con el Síndrome Respiratorio de Oriente Medio (MERS). El 31 de diciembre del año pasado, las autoridades chinas alertaron a la Organización Mundial de la Salud de un brote de una nueva cepa de coronavirus que causaba enfermedades graves, que posteriormente fue nombrada SARS-CoV-2. Hasta el 16 de marzo de 2020, se han documentado casi 167.500 casos de COVID-19, aunque es probable que no hayan sido diagnosticados muchos más casos leves. El virus ha matado a más de 6.600 personas (datos más recientes, del 2 de mayo de 2020, revelan que el COVID-19 ha afectado a 3.496.622 en el mundo, con 244.870 casos mortales, aprox. el 7%)
Poco después de que comenzara la epidemia, unos científicos chinos secuenciaron el genoma del SARS-CoV-2 y pusieron los datos a disposición de los investigadores de todo el mundo. Los datos de la secuencia genómica resultante han demostrado que las autoridades chinas detectaron rápidamente la epidemia y que el número de casos de COVID-19 ha ido aumentando debido a la transmisión de persona a persona después de una sola introducción en la población humana. Andersen y sus colaboradores de otras instituciones utilizaron estos datos de secuenciación para explorar los orígenes y la evolución del SARS-CoV-2 centrándose en varias características reveladoras del virus.
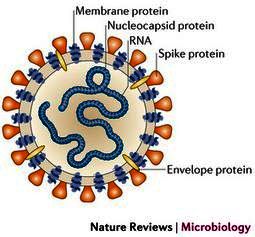 Los científicos analizaron la secuencia genética codificante de la proteína de las espinas, de la armadura exterior del virus, que utiliza para agarrar y penetrar las paredes externas de las células humanas y animales. Más específicamente, se centraron en dos características importantes de la proteína las espinas: el dominio de unión a receptores (RBD), una especie de gancho de agarre a las células huésped, y el sitio de escisión, un abrelatas molecular que permite que el virus abra la pared penetre en las células huésped.
Los científicos analizaron la secuencia genética codificante de la proteína de las espinas, de la armadura exterior del virus, que utiliza para agarrar y penetrar las paredes externas de las células humanas y animales. Más específicamente, se centraron en dos características importantes de la proteína las espinas: el dominio de unión a receptores (RBD), una especie de gancho de agarre a las células huésped, y el sitio de escisión, un abrelatas molecular que permite que el virus abra la pared penetre en las células huésped.
Evidencia de evolución natural
Los científicos encontraron que la porción de RBD de la proteína de la espina del SARS-CoV-2 había evolucionado para dirigirse eficazmente a una característica molecular en el exterior de las células humanas llamada ACE2, un receptor implicado en la regulación de la presión arterial. Si bien su análisis sugirió que el SARS-CoV-2 se puede unir al ACE2 humano con alta afinidad, los análisis computacionales predicen que la interacción no es ideal y que la secuencia de RBD es diferente de las óptimas de la unión a los receptores. que se muestran en otros SARS-CoV.
Esto sugiere que el SARS-CoV-2 es el resultado de una selección natural. Esta evidencia de evolución natural fue apoyada por datos de la estructura molecular general de la columna vertebral del genoma del SARS-CoV-2– . Si alguien estuviera tratando de diseñar un nuevo coronavirus como patógeno, lo habría construido a partir de la columna vertebral de un virus que se sabe que causa enfermedad. Pero los científicos encontraron que la columna vertebral del SARS-CoV-2 difería sustancialmente de las de los coronavirus ya conocidos y en su mayoría se parecía principalmente a la de los virus relacionados que se encuentran en murciélagos y pangolines.
Según Andersen «Estas dos características del virus, las mutaciones en la porción de RBD de la proteína de las espinas y su columna vertebral distinta, descartan la manipulación de laboratorio como un origen potencial para SARS-CoV-2«. Josie Golding, PhD, epidémiólogo líder del Wellcome Trust, con sede en el Reino Unido, dijo que los hallazgos de Andersen y sus colegas son «crucialmente importantes para aportar una visión basada en la evidencia de los rumores que han estado circulando sobre los orígenes del virus (SARS-CoV-2) causando COVID-19«. Golding añade que “El virus es el producto de la evolución natural«, poniendo fin a cualquier especulación sobre la ingeniería genética deliberada.
Posibles orígenes del virus
Basados en su análisis de secuenciación genómica, Andersen y sus colaboradores concluyeron que los orígenes más probables para SARS-CoV-2 siguieron uno de dos escenarios posibles. En un escenario, el virus evolucionó a su estado patógeno actual a través de la selección natural en un huésped no humano y luego saltó a los seres humanos. Así es como han surgido brotes de coronavirus anteriores, con los seres humanos contrayendo el virus después de la exposición directa a civetas (SARS) y camellos (MERS). Los investigadores propusieron murciélagos como el reservorio más probable para SARS-CoV-2 ya que es muy similar a un coronavirus de murciélagos. Sin embargo, no hay casos documentados de transmisión directa de murciélagos a humanos, lo que sugiere que probablemente está involucrado un huésped intermedio entre murciélagos y humanos.
En este escenario, ambas características distintivas de la proteína de las espinas del SARS-CoV-2, la porción de RBD que se une a las células y el sitio de escisión que abre el virus, habrían evolucionado a su estado actual antes de entrar en los seres humanos. En este caso, la epidemia actual probablemente habría surgido rápidamente tan pronto como los seres humanos se infectaron, ya que el virus ya habría evolucionado las características que lo hacen patógeno y capaz de propagarse entre las personas.
En el otro escenario propuesto, una versión no patógena del virus saltó de un huésped animal a los seres humanos y luego evolucionó a su estado patógeno actual dentro de la población humana. Por ejemplo, algunos coronavirus de pangolines, mamíferos similares a armadillos que se encuentran en Asia y África, tienen una estructura de RBD muy similar a la del SARS-Cov2. Un coronavirus de un pangolín podría haber sido transmitido a un humano, ya sea directamente o a través de un huésped intermediario como civetas o hurones.
Entonces la otra proteína de las espinas distintiva característica del SARS-CoV-2, el sitio de escisión, podría haber evolucionado dentro de un huésped humano, posiblemente a través de una circulación no detectada limitada en la población humana antes del comienzo de la epidemia. Los investigadores encontraron que el sitio de escisión SARS-CoV-2, parece similar a los sitios de escisión de cepas de la gripe aviar que se ha demostrado que se transmite fácilmente entre las personas. SARS-CoV-2 podría haber evolucionado un sitio de escisión tan virulento en las células humanas y a partir de ahí arrancó la epidemia actual, ya que el coronavirus posiblemente se habría vuelto mucho más capaz de propagarse entre las personas.
El coautor del estudio Andrew Rambaut advirtió que es difícil, si no imposible, saber en este punto cuál de los escenarios es más probable. Si el SARS-CoV-2 entró en humanos en su forma patógena actual a partir de una fuente animal, aumenta la probabilidad de brotes futuros, ya que la cepa causante de la enfermedad del virus todavía podría estar circulando en la población animal y podría saltar una vez más a los seres humanos. Las posibilidades son menores de que un coronavirus no patógeno entre en la población humana y luego evolucione a las del SARS-CoV-2.
La financiación de la investigación fue proporcionada por los Institutos Nacionales de Salud de los Estados Unidos, los Pew Charitable Trusts, el Wellcome Trust, el Consejo Europeo de Investigación y una beca ARC Australian Laureate.